Trayectoria del virus de la influenza en Yucatán: filogenética y mutaciones
Por Marytere Narváez
Mérida, Yucatán. 8 de septiembre de 2015 (Agencia Informativa Conacyt).- Guadalupe Ayora Talavera, miembro del Sistema Nacional de Investigadores (SNI) nivel I, ha sido pionera en el estudio de la influenza en el país a partir de sus investigaciones en el Laboratorio de Virología del Centro de Investigaciones Regionales Dr. Hideyo Noguchi de la Universidad Autónoma de Yucatán (CIR Biomédicas Uady).
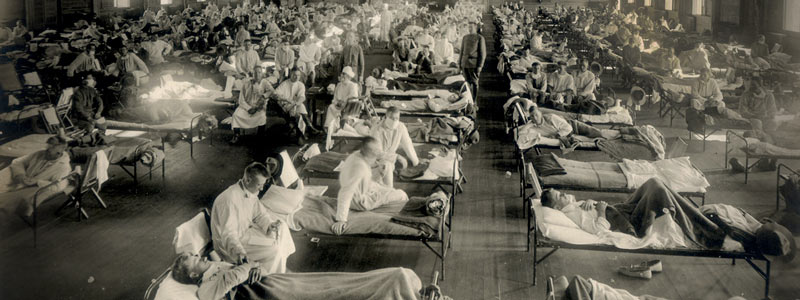 Pandemia de influenza de 1918. Campo Funston, Kansas. Imagen cortesía de National Museum of Health and Medicine, a través de Wikimedia Commons.
Pandemia de influenza de 1918. Campo Funston, Kansas. Imagen cortesía de National Museum of Health and Medicine, a través de Wikimedia Commons.
Según la especialista, la primera huella del virus de la influenza se remonta a textos que datan de la época de Sócrates en el año 412 a. C., donde se relata una epidemia con síntomas de lo que ahora conocemos como gripe. Los registros médicos de la primera pandemia del virus datan de 1918, durante la última etapa de la Primera Guerra Mundial. Fue hasta 1933 que Christopher Andrews y Wilson Smith lograron obtener la caracterización del virus de la influenza y se estudió la conformación de su genoma, las proteínas de la superficie y la respuesta inmune del huésped, de acuerdo con la revista La ciencia y el hombre de la Universidad Veracruzana.
En México, la presencia de la influenza se detectó por primera vez en 1994 y 1995 con el virus aviar subtipo H5N1, que fue controlado y erradicado mediante una campaña de la Secretaría de Agricultura, Ganadería, Desarrollo Rural, Pesca y Alimentación (Sagarpa), pero los estudios sobre la epidemiología eran mínimos y la pérdida económica fue devastadora.
Primeros estudios de la influenza en México
 Guadalupe Ayora.En 1998, Ayora Talavera se integró en un proyecto financiado por Aventis Pasteur y el Instituto de Diagnóstico y Referencia Epidemiológicos (Indre), conocido como Grupo Regional de Estudio de la Gripe, en el que participaban seis estados de la república. Aventis Pasteur financió los reactivos para la realización de diagnósticos y el grupo se encargó de establecer una red de búsqueda de casos de influenza en los hospitales de cada región.
Guadalupe Ayora.En 1998, Ayora Talavera se integró en un proyecto financiado por Aventis Pasteur y el Instituto de Diagnóstico y Referencia Epidemiológicos (Indre), conocido como Grupo Regional de Estudio de la Gripe, en el que participaban seis estados de la república. Aventis Pasteur financió los reactivos para la realización de diagnósticos y el grupo se encargó de establecer una red de búsqueda de casos de influenza en los hospitales de cada región.
En Yucatán se incorporaron los municipios de Tizimín, Valladolid, Progreso y Mérida, donde se colectaron muestras clínicas de pacientes que accedían a participar en el estudio. De esta manera, por primera vez se realizó un reporte del virus de influenza a nivel nacional.
“Todo el trabajo que se hizo durante los primeros ocho a diez años sentó las bases para que se pudiera conocer un poco más de la epidemiología de la influenza en México, pero a pesar de eso no fue hasta que ocurrió la pandemia que se le dio la importancia que tiene a la enfermedad", comentó la doctora.
El 24 de abril de 2009 se conoció la presencia en humanos de un virus desconocido hasta entonces. Se trataba de un virus de influenza A subtipo H1N1 (H1: hemaglutinina, primera de los 18 subtipos que existen; N1: neuraminidasa, primera de los 11 subtipos identificados hasta la fecha) con una conformación procedente de cuatro diferentes líneas filogenéticas que incluían la aviar, la humana, la porcina asiática y la americana.
Barrera entre especies
De acuerdo con la especialista, el virus de influenza A puede transmitirse entre aves, mamíferos y humanos. En algunos casos, el virus viaja de un país a otro utilizando como medio de transporte las aves migratorias.
Una de las características principales del virus es que su material genético ARN está segmentado, de manera que permite que cuando una misma célula es infectada por dos virus simultáneamente, ocurra una combinación de sus segmentos y una reorganización de genes que podría generar los virus pandémicos.
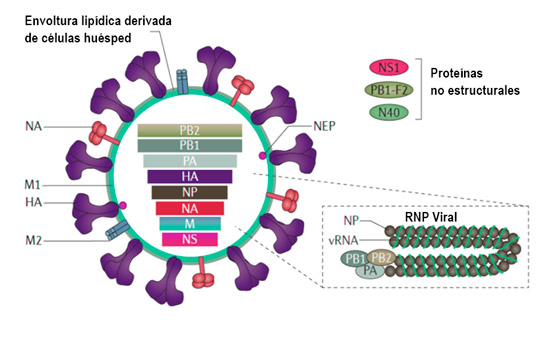 Virus de la influenza.
Virus de la influenza.
Entre humanos, la transmisión es por vía aérea y contacto directo. Las partículas están contenidas en las gotas de aerosol de los fluidos que se expelen al estornudar o toser, por lo que tiene que haber proximidad entre las personas para que las partículas virales puedan ser ingeridas en la respiración y ocurra el contagio. En condiciones de humedad, el virus se mantiene viable y se inicia el proceso de replicación en el momento en que hace contacto con otro individuo.
En el caso de los cerdos y de los humanos, la transmisión ocurre por contacto directo a través de las secreciones nasales de los cerdos. En las aves, el virus no se replica en el tracto respiratorio sino en el tracto intestinal, por lo que es excretado por las heces fecales y es más difícil de que llegue a los seres humanos.
Para que se logre romper la barrera entre humanos y otras especies, el virus debe adquirir la capacidad de poder replicarse en el interior del tracto respiratorio humano. En el caso del virus de aves, usualmente no se replica en el tracto respiratorio superior, que es donde se produce la transmisión por vía directa.
Para adaptarse debe pasar por un proceso evolutivo que requiere que el virus realice modificaciones en su genoma, y durante muchos años se consideró que el cerdo era el intermediario necesario entre las aves y el humano, pero cuando emergió la influenza H5N1 en Asia en 1997, la infección pasó directamente del ave al humano. En la pandemia del año 2009 el cerdo sirvió nuevamente como huésped intermediario en el que se combinaron los genomas que tenían la capacidad de infectar a los humanos.
Árbol filogenético, ¿de dónde vinieron los virus?
Con el apoyo del Fondo Mixto del Conacyt y el gobierno del estado de Yucatán, se desarrolló el proyecto “Vigilancia epidemiológica del virus de influenza H1N1 y detención de variantes genéticas con potencial epidémico en humanos”, cuyo propósito fue realizar una caracterización molecular del virus de influenza de índole pandémica en el estado de Yucatán.
Según la investigadora, los primeros datos considerados en el estudio fueron las tres olas epidémicas ocurridas durante la pandemia. Los primeros casos a nivel nacional se reportaron entre marzo y abril, mientras que en Yucatán hubo un desfase de cinco semanas. La segunda ola ocurrió entre junio y julio en la región sureste, entre los estados de Chiapas, Yucatán, Campeche y Quintana Roo; mientras que en el centro y en el norte del país los registros disminuyeron. La tercera ola epidémica ocurrió nuevamente en el centro y en el norte, siendo incluso más fuerte que la ocurrida al principio del año. En este último caso, en Yucatán los casos fueron mínimos.
"Esa información de cierta manera se relacionaba con algunos estudios previos que ya habíamos observado, pues a diferencia de lo que ocurre en el centro del país, en Yucatán el brote de influenza sucede en la época de lluvia, o sea, entre junio y septiembre", comentó la doctora Ayora Talavera.
 El laboratorio brindó la infraestructura física y diagnóstica a los Servicios de Salud del Estado de Yucatán (SSY), donde se realizó el aislamiento de virus en muestras clínicas. Con este banco se realizó la extracción del material genético del virus y se sometió a un proceso de secuenciación, que consiste en la realización de una copia de la cadena de ARN del gen para saber cuáles son las bases que están contenidas en ella. Para conocer la secuencia contenida en cada cadena se requiere de una enzima polimerasa que traduce los complementos de cada nucleótido y los resultados se observan mediante un equipo especializado.
El laboratorio brindó la infraestructura física y diagnóstica a los Servicios de Salud del Estado de Yucatán (SSY), donde se realizó el aislamiento de virus en muestras clínicas. Con este banco se realizó la extracción del material genético del virus y se sometió a un proceso de secuenciación, que consiste en la realización de una copia de la cadena de ARN del gen para saber cuáles son las bases que están contenidas en ella. Para conocer la secuencia contenida en cada cadena se requiere de una enzima polimerasa que traduce los complementos de cada nucleótido y los resultados se observan mediante un equipo especializado.
"Básicamente nos enfocamos en dos genes, la hemaglutinina (HA) y la neuraminidasa (NA), proteínas que forman parte de la envoltura del virus, y fueron aproximadamente 60 genomas que pudimos secuenciar", señaló la investigadora.
Una vez obtenidas las secuencias se sometieron a un estudio comparativo entre las muestras de México y el resto del mundo, a partir de lo que se construyeron árboles filogenéticos mediante programación especializada que agrupó las secuencias de acuerdo con la distancia que estaban unas de otras.
En el análisis de los genomas de 2009 se reportó la existencia de dos orígenes. El grupo uno provenía de México, Texas y California; mientras que el grupo dos provenía de Nueva York. En este análisis se encontró que los virus de Yucatán provinieron del grupo dos y no del centro del país, como era de esperarse.
En el 2010 se reportaron pocos casos de infección con el virus pandémico, principalmente a principios de año. En 2011 en Yucatán no hubo ningún caso, mientras que en Chihuahua se registró un brote de virus de influenza que provocó casos fatales de infección. En el caso de las muestras colectadas en el 2012, los virus de Yucatán están agrupados en el mismo clado que los virus del resto del país, por lo que puede decirse que tuvieron el mismo origen.
En el caso de las muestras de 2013, se observó que las cepas de Yucatán se encontraban en un grupo completamente distinto al que pertenecían los virus del resto de México, por lo que puede pensarse que el origen del virus en el sureste estuvo asociado a la afluencia turística norteamericana.
Nueva mutación en Yucatán
Con la asesoría de la doctora Ayora Talavera y con el apoyo de una beca mixta del Conacyt, Adriana A. Matos Patrón realizó el doctorado en Ciencias de la  Adriana A. Matos.Salud con el tema “Estudio de la afinidad de unión del virus de la influenza H1N1 al ácido siálico”, con el objetivo de investigar las características de los virus de influenza obtenidos de pacientes de casos severos y fatales de infección en Yucatán durante la pandemia de 2009.
Adriana A. Matos.Salud con el tema “Estudio de la afinidad de unión del virus de la influenza H1N1 al ácido siálico”, con el objetivo de investigar las características de los virus de influenza obtenidos de pacientes de casos severos y fatales de infección en Yucatán durante la pandemia de 2009.
Un aspecto importante de los virus que poseen genoma de ARN es su alta tasa de mutación debido a que durante la replicación son introducidos errores en el genoma a causa de la deficiencia de corrección de la ARN polimerasa. En el caso del virus de influenza las mutaciones introducidas están asociadas principalmente con cambios en las proteínas HA y NA; y pueden ocasionar que los anticuerpos producidos durante la infección no reconozcan las nuevas variantes virales, por lo que es necesario actualizar la vacuna anualmente.
El estudio de la doctora Matos Patrón se centró precisamente en estudiar una región de la proteína HA que se conoce como el sitio de unión al receptor y el efecto de mutaciones en este sitio en la replicación del virus en células epiteliales del tracto respiratorio humano. Estas células se caracterizan por contener una membrana apical con glicoproteínas y glicolípidos (entre otros) que poseen una molécula de ácido siálico que encaja perfectamente con el sitio de unión al receptor de la HA del virus, y es a través del contacto entre ambos que se inicia el proceso unión y entrada a la célula respiratoria.
La molécula de ácido siálico puede estar ligada a la glicoproteína en dos conformaciones diferentes, alfa 2,6 (α2,6) si se encuentra en las células del tracto respiratorio superior, o alfa 2,3 (α2,3) se si encuentra en las células epiteliales de bronquiolos y alveolos.
Una de las razones por las que se había preservado la barrera de especie entre aves y humanos es porque los virus de influenza de origen humano reconocen mayormente el ácido siálico en la conformación α2,6, mientras que los virus de influenza aviar tienen afinidad por el α2,3.
En uno de los 15 casos fatales de infección registrados en Yucatán, se encontró que la muestra clínica del paciente contenía virus con dos mutaciones, una en la posición D225N (cambio de aminoácido ácido aspártico o D por el aminoácido asparagina o N) y otra en la posición S188N (cambio de aminoácido serina o S por el aminoácido asparagina o N).
“Estos dos sitios son muy importantes en el reconocimiento del ácido siálico de la célula huésped por el virus de influenza. Otros estudios han reportado mutaciones en estos sitios de la HA; sin embargo, los cambios de aminoácidos son diferentes y nunca antes se habían detectado simultáneamente como se encontró en este caso fatal de Yucatán", comentó en entrevista la doctora Matos Patrón.
Para estudiar el efecto de las mutaciones halladas, se implementó la metodología de genética reversa, que permite analizar de manera individual y en conjunto las mutaciones encontradas, S188N y D225N.
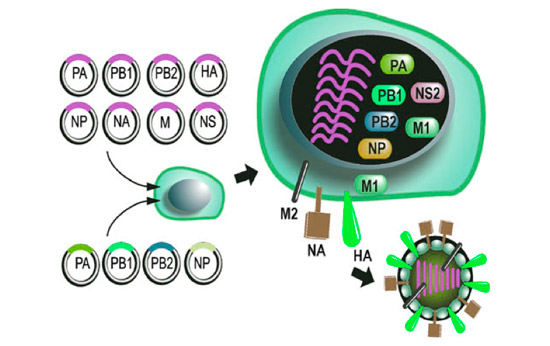 Sistema de genética reversa.
Sistema de genética reversa.
Se clonó cada uno de los ocho segmentos del virus en un vector de expresión, obteniendo un total de ocho plásmidos que se introdujeron en una célula eucariota (proceso mejor conocido como transfección o introducción de ADN a células eucariotas). Haciendo uso de la maquinaria celular y a través de las polimerasas propias del virus, de este proceso de transfección se crearon células de virus de influenza infecciosos.
“La genética reversa vino a revolucionar totalmente el estudio del virus de influenza. Actualmente se está tratando que las vacunas se produzcan a través de esta técnica porque es un proceso más rápido que el que comúnmente se utiliza con los embriones de pollo, y esto nos ha permitido caracterizar cada uno de los genes, cada mutación que encontremos en un virus de influenza”, señaló la doctora Ayora Talavera.
Con las mutaciones S188N y D225N contenidas en HA se generaron cuatro virus diferentes: uno tenía la mutación individual HA-D225N, otro la HA-S188N y uno más que contenía ambas mutaciones HA-D225N + S188N. Las características de replicación y su afinidad por el receptor de ácido siálico de estos virus con las mutaciones se compararon con el cuarto virus generado, virus silvestre por no contener ningún cambio.
Los estudios biológicos se realizaron en cultivos de células epiteliales del tracto respiratorio humano que se caracterizan por contener células ciliadas que expresan principalmente ácido siálico α2,3 y células no ciliadas que expresan principalmente α2,6, lo que hace de este cultivo un sistema biológico perfecto para estudiar las propiedades de afinidad por este tipo de receptor.
A través de los experimentos en estos cultivos de células epiteliales de tracto respiratorio se logró evidenciar que los virus que contenían la mutación en HA D225N se replicaron principalmente en células no ciliadas que expresaban ácido siálico α2,6, aunque la cantidad de virus detectada fue menor en comparación con los otros dos mutantes. De manera similar, los virus con la mutación en HA S188N también infectaron principalmente las células no ciliadas con α2,6; sin embargo, fueron más eficientes en su replicación. En contraste, los virus con la doble mutación S188N+D225N infectaron tanto las células ciliadas como las no ciliadas y, por lo tanto, su replicación fue mucho mayor que los otros dos virus.
Con esto se demostró que cuando el virus tiene la mutación D225N su capacidad de replicación se ve afectada desde el inicio del ciclo al disminuir su afinidad por el ácido siálico α2,6 como consecuencia de la mutación localizada específicamente en el sitio de unión al receptor. Por tanto, el virus se ve forzado a adquirir una mutación adicional, la S188N, que no solo le permitió recuperar su afinidad por el receptor α2,6, sino que además le confirió la capacidad de reconocer el α2,3, facilitando la replicación del virus en el tracto respiratorio superior e inferior del paciente y muy probablemente contribuyendo a la severidad del caso. Estos resultados sugieren una relación directa entre el caso fatal de infección y las dos mutaciones encontradas.
"Lo anterior sienta las bases para continuar el estudio del virus de influenza, así como analizar y evaluar las propiedades biológicas de nuevas variantes de virus que pudieran en algún momento ser de relevancia epidémica”, mencionó Matos Patrón.
Políticas públicas
Para conocer con mayor precisión el carácter macro de la incidencia viral de la influenza, el Laboratorio de Virología desarrolla la propuesta de un proyecto interdisciplinario en conjunto con  la Universidad Nacional Autónoma México (UNAM) y el Instituto Fogarty de Estados Unidos, en el que se incluyen los parámetros ambientales y los datos epidemiológicos de estudios comparativos en Yucatán, Distrito Federal y Nuevo León.
la Universidad Nacional Autónoma México (UNAM) y el Instituto Fogarty de Estados Unidos, en el que se incluyen los parámetros ambientales y los datos epidemiológicos de estudios comparativos en Yucatán, Distrito Federal y Nuevo León.
Asimismo, también se desarrolla una propuesta en conjunto con la Universidad de Florida para fortalecer el sistema de vigilancia permanente de la influenza a través de la incorporación de redes sociales, con el objetivo de conocer lo que sucede con la epidemiología del virus en el estado durante todo el año y mejorar las políticas públicas de vacunación, pues uno de los aspectos más sobresalientes que se han logrado conocer es que en el sureste las epidemias de influenza ocurren de manera diferente que en el centro y el norte del país.
“El programa de vacunación en México se inicia en el mes de octubre y termina en febrero del año siguiente en sincronía con la epidemia anual de influenza. Sin embargo, en Yucatán la epidemia de influenza ocurre entre los meses de junio y septiembre, y por lo tanto la vacunación tendría mejor efecto en la población yucateca si se vacunara en los meses previos a la epidemia (marzo-abril), y esto es parte de lo que considera este proyecto, con el objetivo de favorecer las políticas públicas de salud del estado”, apuntó Ayora Talavera.

Esta obra cuyo autor es Agencia Informativa Conacyt está bajo una licencia de Reconocimiento 4.0 Internacional de Creative Commons.

